Python/PySpark(Spark.ml)のRandomForestClassifierにnumpyのデータを使ってみる
2017-09-15 追記
scikit-learnを使って同じことをする記事も書きました。比べてみると面白いかも?
PySpark使えば簡単に分散環境で機械学習出来て楽しい的な話を聞いたのでやってみました。 結論から言うと、そんなに簡単じゃなかった。…前処理が。
今回は、sklearnのirisをRandomForestでクラスタリングしてみました。 データの用意が簡単なのと、numpy.arrayとspark.dataframeの相互変換の方法が知りたかったので。
とりあえずプロット
ここはPySpark関係無いです。
>>> import matplotlib.pyplot as plt >>> import sklearn.datasets >>> rawdata = sklearn.datasets.load_iris() >>> plt.scatter(rawdata.data[:,0], rawdata.data[:,2], c=[['orange', 'green', 'blue'][x] for x in rawdata.target]) >>> plt.show()
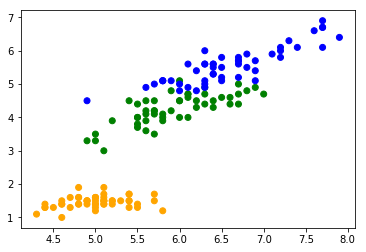
今回は花弁とがくの長さだけ使って幅は使いません。2次元のがプロットしやすいし。
PySparkを使う準備
とてもだいじ。でもわりと省略してるブログとかドキュメントが多くて詰むんですよね…。
>>> import pyspark >>> sc = pyspark.SparkContext.getOrCreate() >>> session = pyspark.sql.SparkSession(sc)
pyspark.SparkContext.getOrCreate()はその名の通り、初回ならコンテキストを作成、そうでなければ既存のものを取得してくるやつです。
一回しか使わないならpyspark.SparkContext()でも良いみたいだけど、とりあえずgetOrCreate()で良さそう。
pyspark.sql.SparkSessionはspark.DataFrameを使うために必要みたいです。
古いブログとかだとpyspark.sql.SQLSessionを使ってたりするけど、今はこっちを使うべきらしい(?)
numpy.ndarray -> pyspark.DataFrame
sklearnで読み込んだnumpy.arrayなデータをSparkのDataFrameってやつに変換します。
DataFrameって言われてpandasの二次元な表を想像してしまったのですが、なんか若干微妙な形のデータフレームなので注意が必要です。かなり悩んだ。
データフレームとしては2列だけで、片方の列にデータのラベルを、もう一つの列に特徴量を全部突っ込むみたいです。
listで言うと、次のような感じ。
>>> data = [['typeA', [1, 2, 3]], ... ['typeA', [1, 2, 4]], ... ['typeB', [2, 3, 4]], ... ...]
中途半端に三次元テーブル? というかスキーマレス? なんて言えば良いのか分からない。
で、実際のデータの作り方は次のように。
>>> import pyspark.ml.linalg >>> data = session.createDataFrame([ ... (int(t), pyspark.ml.linalg.Vectors.dense([d[0], d[2]])) ... for t, d in zip(rawdata.target, rawdata.data) ... ], ['label', 'features']) ... >>> print(data) DataFrame[label: bigint, features: vector]
これで、Spark側からデータセットを扱う準備が出来ました。
訓練用とテスト用のデータを分ける
これは結構簡単に出来てうれしい。
ここでは7:3の割合で分割してみました。
>>> traningData, testData = data.randomSplit([0.7, 0.3])
データの正規化的なアレをするヤツを作る
これが何なのかさっぱり分かっていない。 値域の正規化とかラベルの整理とかしてくれるらしいのだけれど、分かりません。 ちなみに、今回のデータセットだと無くても動きました。
>>> import pyspark.ml.feature >>> labelIndexer = pyspark.ml.feature.StringIndexer(inputCol='label', outputCol='indexedLabel').fit(data) >>> featureIndexer = pyspark.ml.feature.VectorIndexer(inputCol='features', outputCol='indexedFeatures', maxCategories=4).fit(data) >>> print('original =', data) original = DataFrame[label: bigint, features: vector] >>> print('label =', labelIndexer.transform(data)) label = DataFrame[label: bigint, features: vector, indexedLabel: double] >>> print('feature =', featureIndexer.transform(data)) label = DataFrame[label: bigint, features: vector, indexedFeatures: vector]
outputColに指定された名前の新しい列を作って、そこにinputColで指定された列を処理した結果を入れているみたいです。
bigintがdoubleになったりしてるし、何かを良い感じにしてくれているっぽい。
学習器を作って前処理のヤツと繋ぎ合わせる
やっとデータの準備が出来たので、学習器を作ります。あと少し。 先ほどのIndexer二つで前処理を行なって、それからRandomForestClassifierを使って学習を行ないます。
小規模なデータなら一時変数使えば良いじゃんって感じなのですが、分散処理するような大規模データだとそうも行かず。 というわけで、その辺の処理をよしなにやってくれるPipelineというものがあるそうです。なのでこれを使います。
>>> import pyspark.ml >>> import pyspark.ml.classification >>> rf = pyspark.ml.classification.RandomForestClassifier(labelCol='indexedLabel', featuresCol='indexedFeatures', numTrees=10) >>> pipeline = pyspark.ml.Pipeline(stages=[labelIndexer, featureIndexer, rf])
学習して、実行する
作ったPipelineを使ってモデルを作ります。
>>> model = pipeline.fit(traningData)
Pipelineの.fit()を呼ぶと学習結果のモデルが返ってくるので、これを保存します。
scikit-learnのやつと違って、Pipeline自体はモデルを保持しないっぽいので注意が必要です。
で、modelを使ってテストデータを分類してみます。
>>> predictions = model.transform(testData) >>> predictions.select('label', prediction', 'features').show() +-----+----------+---------+ |label|prediction| features| +-----+----------+---------+ | 0| 0.0|[4.7,1.6]| | 0| 0.0|[4.8,1.4]| | 0| 0.0|[4.8,1.6]| | 0| 0.0|[4.8,1.9]| | 0| 0.0|[4.9,1.5]| | 0| 0.0|[5.0,1.5]| | 0| 0.0|[5.1,1.5]| | 0| 0.0|[5.4,1.3]| | 0| 0.0|[5.7,1.7]| | 0| 0.0|[4.4,1.3]| | 0| 0.0|[4.9,1.5]| | 0| 0.0|[5.3,1.5]| | 1| 1.0|[5.6,4.5]| | 1| 2.0|[5.9,4.8]| | 1| 1.0|[6.0,4.0]| | 1| 1.0|[6.1,4.0]| | 1| 1.0|[6.3,4.7]| | 1| 2.0|[6.3,4.9]| | 1| 1.0|[6.5,4.6]| | 1| 1.0|[6.6,4.6]| +-----+----------+---------+ only showing top 20 rows
出来た。やったぜ。(出力は実行する度に変わります)
labelがデータセットの教師データ、predictionがモデルを使って分類した結果です。ちょっと間違ってるけどまあ良い感じ。
精度を確かめる
結果が出たので、今度は精度の評価をしてみます。 自分で計算しても良い気がするけれど、評価用のクラスが用意されているのでこれを使ってみます。
>>> import pyspark.ml.evaluation >>> evaluator = pyspark.ml.evaluation.MulticlassClassificationEvaluator(labelCol='indexedLabel', predictionCol='prediction', metricName='accuracy') >>> accuracy = evaluator.evaluate(predictions) >>> print('accuracy {0:.2%}'.format(accuracy)) accuracy 94.87%
精度は94.87%。良さげ。(出力は実行する度に変わります)
ちょっと長いのでやっぱり自分で計算しちゃっても良い気がするけれど、多分速度とか何かの都合でクラスを使った方が良いのでしょう。たぶん。
ちなみに、自分で計算するとこうなる。
>>> accuracy = predictions[predictions['indexedLabel'] == predictions['prediction']].count() / predictions.count() >>> print('accuracy {0:.2%}'.format(accuracy)) accuracy: 94.87%
pyspark.DataFrame -> numpy.arrary
pyspark.DataFrameの.collect()ってやつを呼ぶと配列になるので、それをnumpy.array()に渡せばnumpyの配列に変換出来ます。
色々経由しててあんまり効率良くなさそうな気がする。
これを使って、どれを間違えたのかをプロットしてみます。
>>> import numpy >>> inputs = numpy.array(predictions.select('features').collect()).reshape(-1, 2) >>> results = numpy.array(predictions.select('label', 'prediction').collect(), dtype=int) >>> plt.scatter(inputs[:,0], inputs[:,1], color=[['orange', 'green', 'blue'][answer] if answer == predict else 'red' for answer, predict in results]) >>> plt.show()
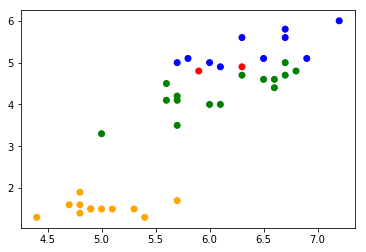
赤い点が間違えて分類したデータ。なるほど間違えそうという感じの場所を間違えている。それっぽい。
まとめ
全部まとめるとこんな感じ。一部端折ってます。
import matplotlib.pyplot as plt import numpy import pyspark import pyspark.ml import pyspark.ml.classification import pyspark.ml.evaluation import pyspark.ml.feature import pyspark.ml.linalg import sklearn.datasets sc = pyspark.SparkContext.getOrCreate() session = pyspark.sql.SparkSession(sc) # データの用意 rawdata = sklearn.datasets.load_iris() data = session.createDataFrame([ (int(t), pyspark.ml.linalg.Vectors.dense([d[0], d[2]])) for t, d in zip(rawdata.target, rawdata.data) ], ['label', 'features']) traningData, testData = data.randomSplit([0.7, 0.3]) # 学習器の組み立て labelIndexer = pyspark.ml.feature.StringIndexer(inputCol='label', outputCol='indexedLabel').fit(data) featureIndexer = pyspark.ml.feature.VectorIndexer(inputCol='features', outputCol='indexedFeatures', maxCategories=4).fit(data) rf = pyspark.ml.classification.RandomForestClassifier(labelCol='indexedLabel', featuresCol='indexedFeatures', numTrees=10) pipeline = pyspark.ml.Pipeline(stages=[labelIndexer, featureIndexer, rf]) # 学習 model = pipeline.fit(traningData) # 評価 predictions = model.transform(testData) evaluator = pyspark.ml.evaluation.MulticlassClassificationEvaluator(labelCol='indexedLabel', predictionCol='prediction', metricName='accuracy') accuracy = evaluator.evaluate(predictions) print('accuracy {0:.2%}'.format(accuracy)) # 結果のプロット inputs = numpy.array(predictions.select('features').collect()).reshape(-1, 2) results = numpy.array(predictions.select('label', 'prediction').collect(), dtype=int) plt.scatter(inputs[:,0], inputs[:,1], color=[['orange', 'green', 'blue'][answer] if answer == predict else 'red' for answer, predict in results]) plt.show()
データの前処理さえ終えてしまえばあとは簡単っぽい。 問題は、前処理の仕方がよく分からない(というかドキュメントが少ない)ことでしょうか。つらい。
参考:
